 n: J! {( t% g) H! v n: J! {( t% g) H! v
中文题目:人类活动对中国沿海鱼类生物多样性的整体影响评价
$ N2 e! M( w% u, `. |" ~6 c 英文题目:Holistic Impact Evaluation of Human Activities on the Coastal Fish Biodiversity in Chinese Coastal Environment
0 }1 d( ^' i" R+ C! u 期刊:Environ. Sci. Technol. 2022
: ^- Y6 @- A3 W: A 第一作者:Wenjun Zhong P9 q, |- `8 ?) G6 k8 |9 _, {+ K
通讯作者:Hongjun Li, Xiaowei Zhang
' J% [; d% _7 G 作者单位:南京大学环境学院;国家海洋环境监测中心
, K5 W4 Q4 s. [! a0 }6 b 方法解读:张政
0 r3 j$ c9 y! _ 正文部分转自公众号【国家海洋环境监测中心】
4 q1 x; O @! z b& e6 V9 s 在此基础上,我们对文章的研究方法进行了详细解读  ) U. r1 A) l0 H. P ) U. r1 A) l0 H. P
正文解读 / r. g/ L- n2 ?" `1 j* W7 l! E* u
环境DNA(Environmental DNA, eDNA)是指从环境样品(如土壤、沉积物、空气、水体等)中提取的DNA,不对任何目标生物进行分离。在海洋生态系统中,通过采集水样和沉积物,收集微型生物的生物个体(个体DNA)和大型生物的碎片或游离DNA(非个体DNA),得到各类海洋生物的环境DNA,从而进行物种或群落的分析。海洋中心李宏俊研究员团队联合南京大学和汕头大学等单位在鸭绿江口和南澳岛海域开展了鱼类环境DNA研究,为评估人类活动对我国近岸鱼类多样性影响提供案例,相关研究成果发表于环境领域顶级期刊Environmental Science & Technology。
$ U u6 u, q9 `* ]! h6 T# e9 V! E- x) Y
随着人类高强度的活动和气候变化,各种人类活动严重威胁近岸海域生态系统质量和稳定性,但是由于缺乏有效的生物多样性监测和评价方法,极大限制了针对不同海洋生态系统环境压力影响的评估。本研究通过整合eDNA技术和生态功能特性,建立了一套评估环境污染和水产养殖对我国近岸海域鱼类生物多样性影响的综合性方法。通过建立我国鱼类12S rRNA条形码参考数据库,提高了eDNA技术对我国海洋鱼类群落分类学注释的准确性,同时分别在序列、分类学和功能尺度计算鱼类群落的多样性,在多个维度提供鱼类群落多样性的分析结果。选择黄海鸭绿江口和南海南澳岛作为代表性研究区域,对本文开发的eDNA生物监测评价工作流程的适用性进行了验证(图1)。
8 i: o) k1 b/ y! X) U  - M+ H1 R S4 G$ O# w' ^! U - M+ H1 R S4 G$ O# w' ^! U
图1 研究框架
4 Z4 w* c- `4 O# O, x 本研究利用便携式水环境DNA过滤仪搭配富集器采集表层海水,通过DNA提取、PCR扩增、DNA文库构建和高通量测序比对等步骤,在鸭绿江口检测到鱼类物种14目24科43属46种,环境DNA所检出鱼类有76.1%可被鸭绿江口历史渔获物种验证,监测结果还表明,处于海洋保护区中的鱼类丰富度显著高于非保护区。南澳岛区域鱼类物种的丰富度是鸭绿江口的1.8倍,南澳岛中低营养级的鱼类的检出率是鸭绿江口地区的1.64倍(图2)。
! m! i5 p$ f, s- Q, F+ x3 v 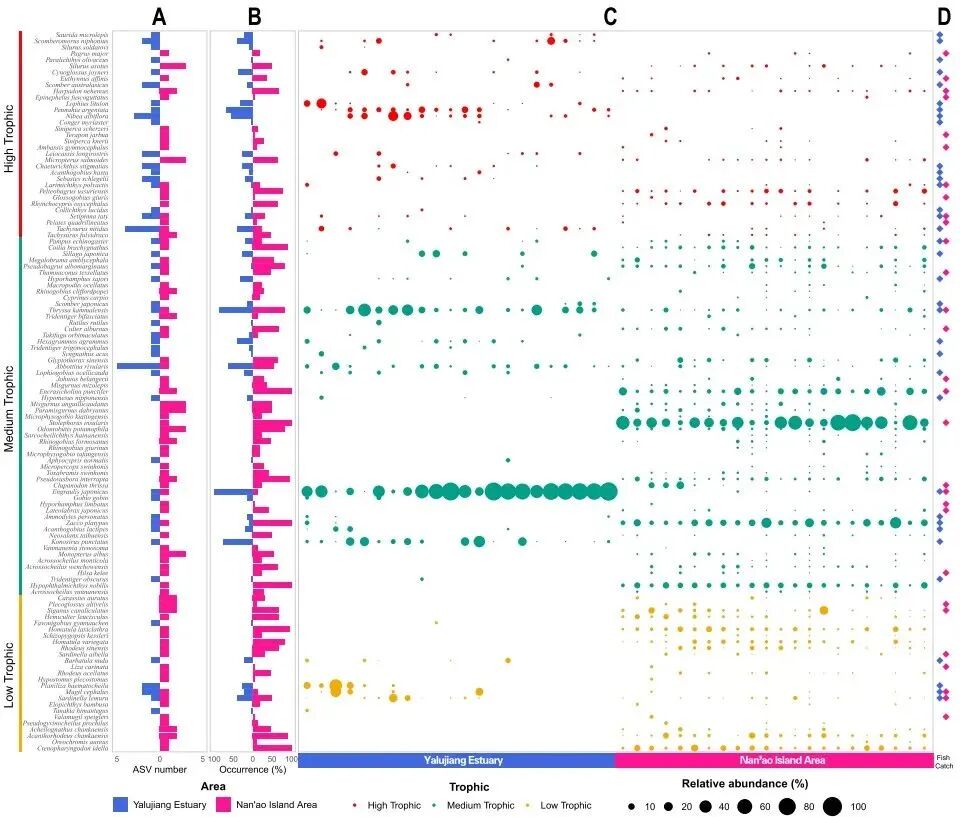
+ ^/ b/ ^* N* S
图2 研究区域不同营养级鱼类物种多样性
' q+ ?6 S9 F7 S2 F; n! s5 l
' n3 n) @* P; B' ?3 P/ S 随着采样站位与岸边的距离逐渐增加,鸭绿江口地区鱼类序列多样性首先快速升高随后保持稳定,而南澳岛地区鱼类序列多样性呈现持续升高趋势。鱼类分类学多样性和功能多样性在鸭绿江口随着离岸距离的增加逐渐降低,而在南澳岛地区,其呈现先增加后降低之后再增加的变化趋势(图3)。此外,无论是在南澳岛还是鸭绿江口地区,鱼类分类学多样性和功能多样性具有显著相关的一致性变化趋势。在鸭绿江口地区,河口污染因素(COD和Zn)导致鱼类群落序列多样性明显下降。在南澳岛地区,养殖区域的鱼类群落序列多样性要低于非养殖区域(图4)。 : F2 Q1 v. @4 v4 V3 n. E d
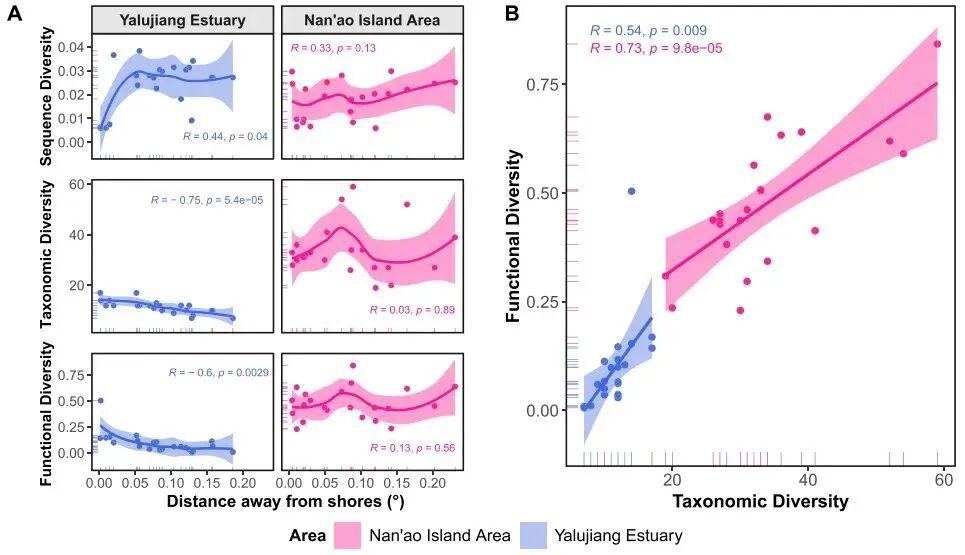
9 ~2 \, A4 U* |1 v ^8 o1 Z; h 图3 研究区域鱼类多样性空间分布规律
0 ?5 [2 Z6 }; p& N, M j* d 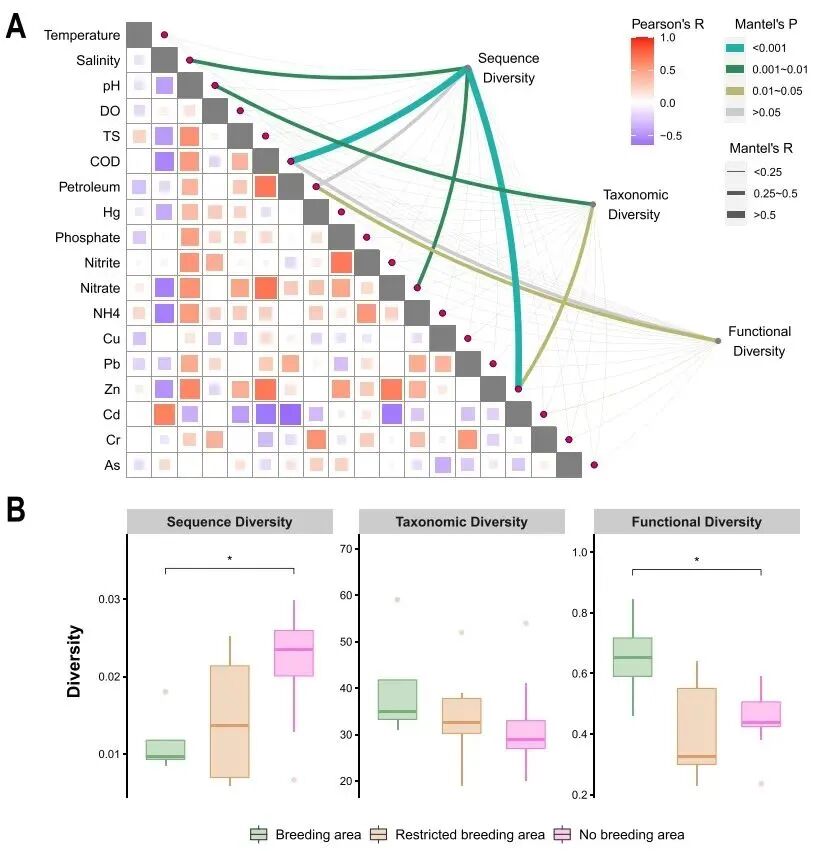
, V/ G- @. E3 l( H& A+ x, V7 K 图4 人类活动对研究区域鱼类多样性影响机制 8 v* f/ e$ h T6 i
本研究认为,目前eDNA研究海洋生物多样性,不能局限在注释到的具体物种,eDNA也不可能检测出来所有的物种。首先参考数据库目前还并不完整,有很多物种目前还没有对应的barcode序列,参考数据库中没有序列就可能检测不到;其次是无论选择哪一段目标序列,都无法做到准确区分所有物种,肯定会存在一些在测序区域序列相似度特别高甚至序列一致而无法得到有效区分的物种。构建针对目标研究区域的个性化参考数据库可能会部分解决注释错误的问题,但是肯定也没办法做到100%的准确,就像本研究专门建立了一个我国鱼类12S rRNA的参考数据库,确实能够提高分类学注释的准确性,但是从结果中也可以看出,eDNA结果和历史观测数据的一致性在鸭绿江口海域为76.09%,但是在南澳岛海域也仅有35.8%。
8 Q' P, S& u. V* ~( A3 p 因此,在进行大型生物eDNA的研究中,应该采取弱化具体物种的研究思路,使用一些综合性的评价指数,比如alpha多样性指数、beta多样性指数、功能多样性,或者是一些生态功能分类,比如营养级、生活习性、食性等,来代替具体的物种以达到研究目的。
$ u1 G: Z7 ]+ K, U- K- \ 下一步,课题组将开展我国近海环境DNA“生命之树”扫描式监测,为摸清我国海洋生物多样性本底提供基础数据,开展不同生物类群生态响应和阈值研究,为构建我国近海生态质量评价标准和业务化监测提供科学依据。  : R" } P6 R7 z& M! {8 v
: R" } P6 R7 z& M! {8 v 方法详解 - b d& b5 B' \/ [
1. 对引物Teleo进行in silico评价
" n' z0 P3 p. c( K (1)从EMBL下载序列信息 # t9 M8 d. J6 b8 T& |
(2)使用引物Teleo针对下载并整合分类信息的序列进行in silico分析,只保留可以被该引物比对出的鱼类信息
9 S8 X( Z0 Y! [ N1 ?- ]: z: [! c (3)分析这些鱼类的世界和中国海域的分布情况,从而说明该引物在中国沿海地区的鱼类检测效力
% g, @& j' \% v' Q 2. 构建中国鱼类12S-DNA条形码参考数据库 % T4 [' {: B& T; M* }6 ^9 F
(1)从fishbase.org获得鱼类分类信息
, |1 r8 L! X1 v9 i$ \ j; a' Z (2)根据分类信息,从NCBI Taxonomy Browser获得每种鱼类的“taxid” ; M3 k s2 f9 S
(3)根据“taxid”搜索获得每种鱼的12S-rDNA序列
. [6 p* t8 D0 A c6 B (4) 如果某鱼种没有公开的12S-rDNA序列但有全基因组信息,从该鱼种的全基因组中选择相应的12S-rDNA;如果一个物种有2个及以上的12S序列时,比较后去重复
9 h" h) n/ X1 h+ U$ d 3. 获得中国鱼类性状数据 # n; {# U) n x* ` Z" g
(1)从fishbase.org 获得迁移类型、摄食类型和营养级指数等性状信息;在 globalbioticinteractions.org上筛选鱼类捕食关系,并将其映射为鱼类食物网 , Y" U# t6 h! s4 e5 g \
(2)从rfishbase 获得洄游等信息 7 Q4 U! X8 c" u4 r4 `) c7 D' a
(3)从fishbase.org 下载具有最大体长数据的整条成鱼的高质量图片和科学侧视图,然后是针对体型变量使用对侧视图图片进行评估。 ; g3 D# o! D& Y5 e8 h( \- I
4. eDNA取样、扩增和测序 ! S$ A$ O3 b6 B& @4 S4 O+ k
(1)在鸭绿江口和南澳岛海域收集海水,过膜 ' I# h5 e$ t: j, W3 }' i- p
(2)使用DNeasy Blood & Tissue Kit提取DNA(66个海水样品、3个空白滤膜、3个提取空白和3个阴性对照) & Y1 Y$ M m1 k0 E% `
(3)使用12S-rRNA引物Teleo扩增 7 U0 u* O* E& {: Q
(4)使用Ion Torrent S5测序仪进行测序 0 h: e( e2 v6 n6 X
5. 生物信息学分析和分类注释
" g* [, A4 D. y7 E7 A1 N0 e, D (1)针对测序数据使用USEARCH的--unoise3命令生成ASV + S7 S' e: e8 `& q( b" q2 O
(2)只保留reads大于10的ASV,并只保留注释到种水平的ASV
* ?7 h% S \+ I! q) o6 p (3)只有在每个站点至少2个重复中出现的ASV才被认作存在,并且对该站点的重复进行平均后四舍五入来合并该站点的ASV reads
6 {% r9 j- e9 T7 |% Q$ e) ` 6. 序列、分类和功能多样性的计算 7 {7 E+ m3 W# n
(1)对特定区域内同一物种的所有ASV序列进行多序列比对,仅保留重叠率>50%的序列 0 X% \7 {& m# |& N
(2)通过跨地区重叠物种的ASV数量和物种内的平均序列变异来进行序列多样性的比较 % f2 d/ ^7 D( Z4 I% L! |; B9 l8 I
(3)根据 计算核苷酸多样性 计算核苷酸多样性
1 H5 {5 y' h% i (4)mFD package计算每个采样点的functional richness (FRic) - F3 v7 n R/ Q
7. 比较区域之间和区域内的多样性变化 6 l8 B# u/ Q( x- c$ C) X' M
(1)序列多样性差异进行Wilcoxon秩和检验
9 }$ u1 y" U9 l* ^+ Q, S (2)β多样性比较 . x! a$ s3 f# C! ~& j
8. 针对不同养殖区域评价河口污染对海岛养殖的影响 " f+ [+ f. ~" H' A# A- d& L
(1)Mantel检验用于确定与鸭绿江口不同鱼类多样性方面显著相关的环境因素 $ ^# h' j; S/ I- ?( f
(2)采用Wilcoxon秩和检验对南澳海域3类养殖区的序列、分类和功能多样性进行检验 (Zhong et al., 2022)  - ?3 ?4 E% n% B5 Z. M# o' G) ^; r - ?3 ?4 E% n% B5 Z. M# o' G) ^; r
参考文献
- D' t% U6 _- u, \ 参考文献: & S8 r5 t, ?6 ~
Zhong, W., Zhang, J., Wang, Z., Lin, J., Huang, X., Liu, W., ... Zhang, X., 2022. Holistic Impact Evaluation of Human Activities on the Coastal Fish Biodiversity in the Chinese Coastal Environment. Environ Sci Technol. " A! e8 y- N7 {+ k
原文链接: 0 l, _, @8 i3 {4 a
https://pubs.acs.org/doi/10.1021/acs.est.2c01339#
& F4 Q+ w- Y9 ~% x: A( m9 f  
* `6 h6 F) l1 ?- s
中国科学院生态环境研究中心
9 _- E6 j- v( }0 K, M. h% U2 @! B- X9 X6 }" G/ E
环境生物技术重点实验室
) x. ]3 Q. X5 ]- B1 O3 n 邓晔 研究员课题组发布  ) a- j6 w. ^- I# C) L3 e; J
) a- j6 w. ^- I# C) L3 e; J 公众号:
" h5 H# e, H. Y( ]( W# u( q 微生态笔记 0 c2 m' W1 |. n' T4 \& g' l+ a
微信号:
( j$ [7 A" `$ r) Y mem_denglab
& P" A D( I, g" N ● 扫码关注我们 - {; E8 v6 L. A& p) \# i# F
# N# r8 W: ~, z- O- P! \4 Q7 ` j& |* s3 W/ q
/ C7 O" O) s. `* y6 u5 p$ K! `% C7 d0 z9 c5 \# ]( W) {+ G
| 

